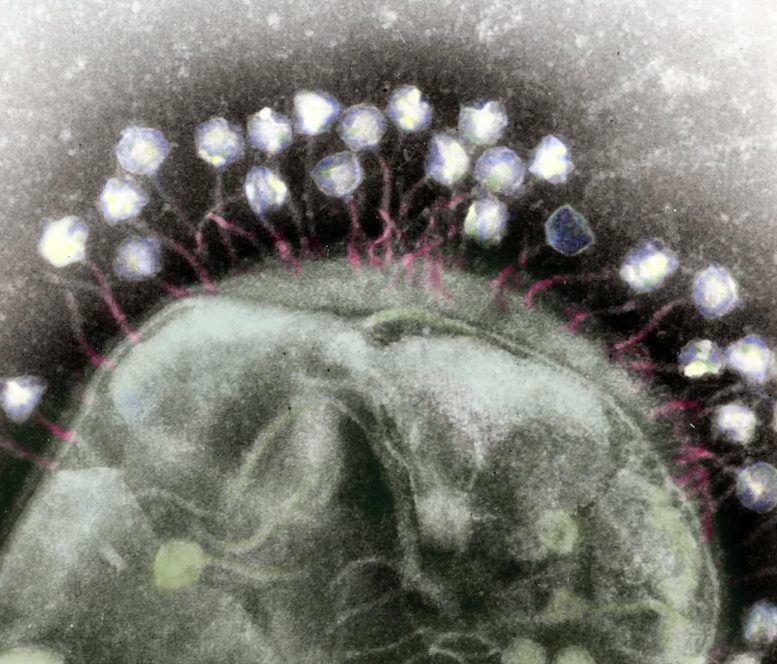
Ein Falschfarbenübertragungselektronenmikrograph mehrerer Bakteriophagen, die an einer bakteriellen Zellwand befestigt sind. Credit: Wikimedia Commons
Ein neuer genetischer Ansatz kann die Untersuchung von Phagen-Mikroben-Interaktionen mit Auswirkungen auf Gesundheit, Landwirtschaft und Klima beschleunigen.
Wissenschaftler suchen ständig nach neuen und verbesserten Möglichkeiten, mit Bakterien umzugehen, sei es, um krankheitserregende Stämme zu eliminieren oder potenziell nützliche Stämme zu modifizieren., Und trotz der zahlreichen cleveren Medikamente und gentechnischen Werkzeuge, die Menschen für diese Aufgaben erfunden haben, können diese Ansätze im Vergleich zu den fein abgestimmten Angriffen von Phagen – den Viren, die Bakterien infizieren-ungeschickt erscheinen.
Phagen entwickeln sich wie andere Parasiten ständig weiter, um ihren spezifischen Bakterienstamm des Wirts anzuvisieren und auszunutzen, und die Bakterien entwickeln sich wiederum ständig weiter, um den Phagen auszuweichen., Diese ewigen Kämpfe um das Überleben ergeben unglaublich unterschiedliche molekulare Arsenale, die Forscher nur schwer untersuchen können, aber dies kann mühsam und arbeitsintensiv sein.
Um Einblicke in diese Verteidigungsstrategien zu erhalten, hat ein Team unter der Leitung von Wissenschaftlern des Berkeley Lab gerade eine effiziente und kostengünstige neue Methode entwickelt. Wie in PLOS Biology berichtet, zeigte das Team, dass eine Kombination von drei Techniken zeigen kann, welche bakteriellen Rezeptoren Phagen nutzen, um die Zelle zu infizieren, sowie welche zellulären Mechanismen die Bakterien verwenden, um auf eine Phagen-Infektion zu reagieren.,
„Trotz fast einem Jahrhundert molekularer Arbeit sind die zugrunde liegenden Mechanismen der Phagen-Wirt-Wechselwirkungen nur für wenige Paare bekannt, bei denen der Wirt ein gut untersuchter Modellorganismus ist, der in einem Labor kultiviert werden kann“, sagte der korrespondierende Autor Vivek Mutalik, ein Wissenschaftler in Berkeley Lab Abteilung für Umweltgenomik und Systembiologie (EGSB). „Phagen stellen jedoch die am häufigsten vorkommenden biologischen Einheiten auf der Erde dar und sind aufgrund ihrer Auswirkungen auf Bakterien wichtige Treiber für den Nährstoffkreislauf der Umwelt, die landwirtschaftliche Produktion sowie die Gesundheit von Mensch und Tier., Es ist unerlässlich geworden, grundlegendere Kenntnisse über diese Wechselwirkungen zu erlangen, um die Mikrobiome des Planeten besser zu verstehen und neue Medikamente wie bakterienbasierte Impfstoffe oder Phagencocktails zur Behandlung von antibiotikaresistenten Infektionen zu entwickeln.“
Licht auf „dunkle Materie“
Der dreipolige Ansatz des Teams, der als barcodierte Bibliotheken für Funktionsverlust und Funktionsgewinn bezeichnet wird, verwendet die etablierte Technik, um Genlöschungen zu erstellen und auch die Genexpression zu erhöhen, um zu identifizieren, welche Gene die Bakterien verwenden, um den Phagen auszuweichen., Diese Information teilt den Wissenschaftlern auch mit, auf welche Rezeptoren die Phagen abzielen, ohne die Genome der Phagen analysieren zu müssen. (Die Wissenschaftler planen jedoch, die Technik für den Einsatz bei Viren in der Zukunft anzupassen, um noch mehr über ihre Funktion zu erfahren.)
Mutalik und seine Kollegen testeten ihre Methode an zwei E. coli-Stämmen, von denen bekannt ist, dass sie auf 14 genetisch unterschiedliche Phagen abzielen., Ihre Ergebnisse bestätigten, dass die Methode reibungslos funktioniert, indem sie schnell die gleiche Reihe von Phagen-Rezeptoren aufdeckten, die zuvor durch jahrzehntelange Forschung identifiziert worden waren, und auch neue Treffer lieferten, die in früheren Studien verpasst wurden.
Eine Künstlerische Umsetzung von Phagen. Credit: Antara Mutalik
Laut Mutalik kann der Ansatz auch skaliert werden, um gleichzeitig Phagen-Beziehungen für Hunderte von Bakterien aus verschiedenen Umgebungen zu bewerten., Dies wird es Wissenschaftlern viel einfacher machen, die biologische „dunkle Materie“ des Planeten zu untersuchen, die sich auf die unkultivierbaren und daher schlecht verstandenen Mikroorganismen bezieht, die in vielen Umgebungen im Überfluss vorhanden sind. Schätzungen zufolge können 99% aller lebenden Mikroorganismen nicht in einem Labor kultiviert werden.
Der Ansatz des Teams bietet auch die Möglichkeit, die in der Phagen-Forschung verwendeten genetischen Ressourcen zu standardisieren, was seit jeher ein Ad-hoc-und hochvariabler Prozess ist, und gemeinsam nutzbare Reagenzien und Datensätze zu erstellen.,
„Die Rolle der Phagen ist ein riesiges ‚bekanntes-Unbekanntes‘, da wir wissen, dass es überall Phagen gibt, aber kaum etwas mehr wissen. Zum Beispiel verstehen wir weniger als 10% der Gene, die in zuvor sequenzierten Phagen-Genomen kodiert sind“, sagte Mutalik. „Jetzt, da wir endlich ein optimiertes Tool zum Betrachten von Phagen haben, gibt es viele aufregende Fragen, die wir beantworten können, und die Möglichkeit, einen Unterschied in der Welt zu machen.,“
Diese Arbeit wurde von Mutalik und anderen EGSB-Wissenschaftlern Adam Arkin und Adam Deutschbauer am Berkeley Lab in Zusammenarbeit mit Forschern der UC Berkeley und des Evergreen State College geleitet. Die Forschung wurde vom Mikrobiologieprogramm des Innovative Genomics Institute und von ENIGMA, einem wissenschaftlichen Schwerpunktprogramm, das vom Berkeley Lab geleitet und vom DOE Office of Science unterstützt wird, finanziert.















