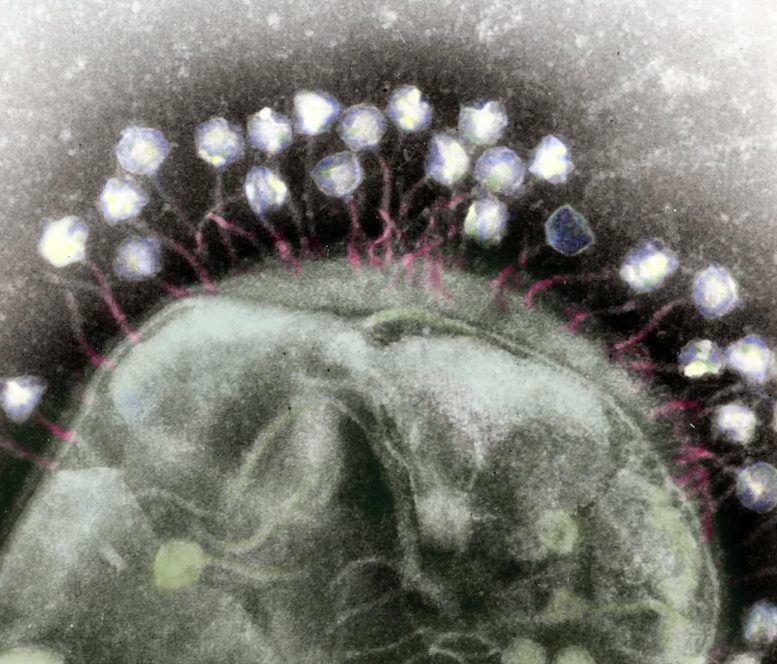
micrographie électronique de transmission en fausse couleur de plusieurs bactériophages attachés à une paroi cellulaire bactérienne. Crédit: Wikimedia Commons
Une nouvelle approche génétique peut accélérer l’étude des interactions phage-microbe avec des implications pour la santé, l’agriculture et le climat.
les scientifiques sont continuellement à la recherche de moyens nouveaux et améliorés de traiter les bactéries, que ce soit pour éliminer les souches pathogènes ou pour modifier les souches potentiellement bénéfiques., Et malgré les nombreux médicaments intelligents et les outils de génie génétique que les humains ont inventés pour ces tâches, ces approches peuvent sembler maladroites par rapport aux attaques finement réglées menées par les phages – les virus qui infectent les bactéries.
les Phages, comme d’autres parasites, sont en constante évolution des moyens de cibler et d’exploiter leur souche bactérienne hôte spécifique, et à leur tour, les bactéries sont en constante évolution des moyens d’échapper aux phages., Ces batailles perpétuelles pour la survie produisent des arsenaux moléculaires incroyablement divers que les chercheurs ont hâte d’étudier, mais cela peut être fastidieux et fastidieux.
pour mieux comprendre ces stratégies défensives, une équipe dirigée par des scientifiques du Berkeley Lab vient de développer une nouvelle méthode efficace et peu coûteuse. Comme indiqué dans PLOS Biology, l’équipe a montré qu’une combinaison de trois techniques peut révéler quels récepteurs bactériens les phages exploitent pour infecter la cellule, ainsi que les mécanismes cellulaires que les bactéries utilisent pour répondre à une infection par phage.,
« malgré près d’un siècle de travail moléculaire, les mécanismes sous-jacents des interactions phage-hôte ne sont connus que pour quelques paires, où l’hôte est un organisme modèle bien étudié qui peut être cultivé en laboratoire”, a déclaré L’auteur correspondant Vivek Mutalik, chercheur à la Division de génomique environnementale et de biologie des systèmes (EGSB) du Berkeley Lab. « Cependant, les phages représentent les entités biologiques les plus abondantes sur Terre et, en raison de leur impact sur les bactéries, ils sont les principaux moteurs des cycles nutritifs de l’environnement, de la production agricole et de la santé humaine et animale., Il est devenu impératif d’acquérir des connaissances plus fondamentales sur ces interactions afin de mieux comprendre les microbiomes de la planète et de développer de nouveaux médicaments, tels que des vaccins à base de bactéries ou des cocktails de phages pour traiter les infections résistantes aux antibiotiques. »
faire la lumière sur la”matière noire »
l’approche à trois volets de L’équipe, appelée bibliothèques de perte de fonction et de gain de fonction, utilise la technique établie de création de suppressions de gènes et d’augmentation de l’expression des gènes pour identifier les gènes utilisés par les bactéries pour échapper aux phages., Ces informations indiquent également aux scientifiques quels récepteurs les phages ciblent sans avoir à analyser les génomes des phages. (Cependant, les scientifiques prévoient d’adapter la technique pour une utilisation sur les virus à l’avenir, pour en apprendre encore plus sur leur fonction.)
Mutalik et ses collègues ont testé leur méthode sur deux souches D’E. coli qui sont connues pour être ciblées par 14 phages génétiquement divers., Leurs résultats ont confirmé que la méthode fonctionne bien en révélant rapidement la même suite de récepteurs de phages qui avaient été précédemment identifiés grâce à des décennies de recherche, et a également fourni de nouveaux succès qui ont été manqués dans des études antérieures.
Un rendu artistique de phages. Crédit: Antara Mutalik
selon Mutalik, L’approche peut également être mise à l’échelle pour évaluer simultanément les relations phagiques pour des centaines de bactéries échantillonnées dans divers environnements., Il sera ainsi beaucoup plus facile pour les scientifiques d’étudier la « matière noire” biologique de la planète, qui fait référence aux micro-organismes inculturables et donc mal compris qui abondent dans de nombreux environnements. En fait, on estime que 99% de tous les micro-organismes vivants ne peuvent pas être cultivés en laboratoire.
l’approche de L’équipe représente également une occasion de normaliser les ressources génétiques utilisées dans la recherche sur les phages, qui a toujours été un processus ad hoc et très variable, et de créer des réactifs et des ensembles de données partageables.,
« le rôle des phages est un énorme « connu-inconnu », car nous savons qu’il y a des phages partout, mais nous ne savons presque rien de plus. Par exemple, nous comprenons moins de 10% des gènes codés dans les génomes de phages précédemment séquencés”, a déclaré Mutalik. « Maintenant que nous avons enfin un outil simplifié pour examiner les phages, il y a beaucoup de questions passionnantes auxquelles nous pouvons commencer à répondre et une opportunité de faire une différence dans le monde., »
Ce travail a été dirigé par Mutalik et ses collègues scientifiques de L’EGSB Adam Arkin et Adam Deutschbauer au Berkeley Lab, en collaboration avec des chercheurs de L’UC Berkeley et de L’Evergreen State College. La recherche a été financée par le programme de microbiologie de L’Innovative Genomics Institute et par ENIGMA, un programme scientifique dirigé par Berkeley Lab et soutenu par le bureau des sciences du DOE.















